دراسة الترابط الجينومي الكامل
دراسة الترابط الجينومي الكامل (اختصاراً GWAS) (ملاحظة 1) هي دراسة تجرى على تعدد أشكال النوكليوتيدات المفردة في كامل الجينوم من أجل ملاحظة صلة الوصل بين التغير الجيني وبين ظهور خصلة أو سمة معينة، وخاصة في مجال الأمراض.
يقوم هذا النوع من الدراسات بفحص كامل الجينوم، وذلك على العكس من أساليب دراسة أخرى، والتي تعتمد على ترشيح جينات معينة للدراسة بناءً على معرفة مسبقة، مما يمكن من التركيز على مناطق جينية محددة. يساعد أسلوب دراسة الترابط الجينومي الكامل على تحديد المتغيرات المترابطة بمرض ما، ولكنه غير قادر من جهة أخرى على تحديد الجين المسبب للسمة المدروسة.[1][2][3]
خلفية
يختلف أي جينوم بشري بملايين الطرق المختلفة. هناك اختلافات صغيرة في النيوكليوتيدات الفردية للجينوم (تعدد أشكال النوكليوتيدات المفردة) بالإضافة إلى العديد من الاختلافات الأكبر، مثل الحذف والإدخال وتغيرات رقم النسخ. قد يسبب أي من هذه التغييرات في سمات الفرد، أو النمط الظاهري، والتي يمكن أن تكون أي شيء من خطر المرض إلى الخصائص الفيزيائية مثل الطول.
في حوالي عام 2000، قبل إدخال دراسات الترابط الجينومي الكامل، كانت الطريقة الأساسية للتحقيق من خلال دراسات الوراثة للارتباط الجيني في العائلات. وقد ثبت أن هذا النهج مفيد للغاية تجاه اضطرابات الجين الواحد. ومع ذلك، بالنسبة للأمراض الشائعة والمعقدة، فقد ثبت صعوبة تكاثر نتائج دراسات الارتباط الجيني. كان البديل المقترح لدراسات الارتباط هو دراسة الارتباط الجيني. يسأل هذا النوع من الدراسة عما إذا عُثِر على أليل المتغير الجيني في كثير من الأحيان أكثر من المتوقع في الأفراد الذين لديهم النمط الظاهري محل الاهتمام (على سبيل المثال مع المرض قيد الدراسة). أشارت الحسابات المبكرة للقوة الإحصائية إلى أن هذا النهج يمكن أن يكون أفضل من دراسات الارتباط في الكشف عن الآثار الوراثية الضعيفة. [4][5][6][7]
بالإضافة إلى الإطار المفاهيمي، مكّنت عدة عوامل إضافية من إجراء دراسات الترابط الجينومي الكامل. كان أحدها ظهور البنوك الحيوية، وهي مستودعات للمواد الجينية البشرية التي قللت بشكل كبير من تكلفة وصعوبة جمع أعداد كافية من العينات البيولوجية للدراسة.[8] وكان مشروع آخر هو مشروع هاب ماب الدولي، الذي حدد منذ عام 2003 غالبية أشكال النيوكلوتايد المشتركة التي تم استجوابها في دراسة الترابط الجينومي الكامل.[9] كما سمح النمط الفرداني الذي حدده مشروع هاب ماب بالتركيز على المجموعة الفرعية من تعدد أشكال النوكليوتيدات المفردة التي من شأنها أن تصف معظم التباين. كما كان تطوير طرق التركيب الوراثي لكل هذه الأشكال المتعددة الأشكال باستخدام مصفوفات التنميط الجيني شرطًا أساسيًا مهمًا. [10]
النظام
النهج الأكثر شيوعًا لدراسات الترابط الجينومي الكامل هو دراسة الحالات والشواهد، الذي يُقارِن بين مجموعتين كبيرتين من الأفراد، مجموعة مراقبة صحية ومجموعة حالة واحدة متأثرة بمرض. جميع الأفراد في كل مجموعة مصنفة وراثيًا لغالبية تعدد الأشكال الشائعة المعروفة. يعتمد العدد الدقيق للنوكليوتيدات المفردة متعددة الأشكال على تقنية التنميط الجيني، ولكن عادةً ما يكون مليون أو أكثر.[11] لكل من هذه النيوكلوتايد يُفحَص ما إذا كان تردد الأليل قد تغير بشكل كبير بين الحالة ومجموعة التحكم. في مثل هذه الإعدادات، فإن الوحدة الأساسية للإبلاغ عن أحجام التأثير هي نسبة الأرجحية. نسبة الأرجحية هي نسبة احتمالين، والتي في سياق دراسات الترابط الجينومي الكامل هي احتمالات الحالة للأفراد الذين لديهم أليل معين واحتمالات الحالة للأفراد الذين ليس لديهم نفس الأليل.
على سبيل المثال، افترض أن هناك أليلين، T و C، عدد الأفراد في مجموعة الحالة التي لديها أليل T يمثله "A" وعدد الأفراد في المجموعة الضابطة التي تحتوي على الأليل T يمثله "B". وبالمثل، فإن عدد الأفراد في مجموعة الحالة التي تحتوي على الأليل C يتم تمثيله بـ "X" وعدد الأفراد في المجموعة الضابطة الذين لديهم الأليل C يمثله "Y". في هذه الحالة، تكون نسبة الأرجحية للأليل T هي A:B (بمعنى "A إلى B"، في مصطلحات الاحتمالات القياسية) مقسومة على X:Y، والتي في التدوين الرياضي هي ببساطة (A/B) / (X/Y).
عندما يكون تردد الأليل في مجموعة الحالة أعلى بكثير منه في المجموعة الضابطة، تكون نسبة الأرجحية أعلى من 1، والعكس صحيح بالنسبة لتردد الأليل المنخفض. بالإضافة إلى ذلك، تُحسَب عادةً قيمة P لأهمية نسبة الأرجحية باستخدام اختبار مربع كاي البسيط. الهدف من دراسة الترابط الجينومي الكامل هو إيجاد نسب رجحان تختلف اختلافًا كبيرًا عن 1 لأن هذا يوضح أن النوكليوتيدات المفردة متعددة الأشكال مرتبط بالمرض. نظرًا لأنه يحصل اختبار العديد من المتغيرات، فمن الممارسات القياسية أن تتطلب قيمة p أقل من 5×10−8 لاعتبار المتغير مهمًا.
هناك العديد من الاختلافات في نهج التحكم في الحالات هذا. بديل شائع لدراسات الترابط الجينومي الكامل وضبط الحالات هو تحليل البيانات المظهرية الكمية، على سبيل المثال الارتفاع أو تركيزات العلامات الحيوية أو حتى التعبير الجيني. وبالمثل، يمكن استخدام إحصاءات بديلة مصممة للهيمنة أو لأنماط الاختراق المتنحي.[12] تتم الحسابات عادةً باستخدام برمجيات المعلوماتية الحيوية مثل SNPTEST و PLINK، والتي تتضمن أيضًا دعمًا للعديد من هذه الإحصائيات البديلة.[13][14] تركز دراسات الترابط الجينومي الكامل على تأثير تعدد أشكال النيوكلوتايد المفردة. ومع ذلك، فمن الممكن أيضًا أن التفاعلات المعقدة بين اثنين أو أكثر من تعدد أشكال النوكليوتيدات المفردة (epistasis) قد تساهم في أمراض معقدة. نظرًا للعدد الأسي المحتمل للتفاعلات، فإن اكتشاف التفاعلات المهمة إحصائيًا في بيانات دراسات الترابط الجينومي الكامل يمثل تحديًا حسابيًا وإحصائيًا. تم تناول هذه المهمة في المنشورات الحالية التي تستخدم الخوارزميات المستوحاة من التنقيب عن البيانات.[15] علاوة على ذلك، يحاول الباحثون دمج بيانات دراسة الترابط الجينومي الكامل مع البيانات البيولوجية الأخرى مثل شبكة تآثرات البروتين-بروتين لاستخراج المزيد من النتائج المفيدة. [16][17]
تتمثل الخطوة الرئيسية في غالبية دراسات الترابط الجينومي الكامل في احتساب الأنماط الجينية عند تعدد الأشكال وليس على شريحة النمط الجيني المستخدمة في الدراسة.[18] تزيد هذه العملية بشكل كبير من عدد النيوكلوتايد التي يمكن اختبارها من أجل الارتباط، وتزيد من قوة الدراسة، وتسهل التحليل التلوي لدراسات الترابط الجينومي الكامل عبر مجموعات متميزة. يُنفَّذ احتساب النمط الوراثي من خلال الطرق الإحصائية التي تجمع بين بيانات دراسات الترابط الجينومي الكامل مع لوحة مرجعية للأنماط الفردانية. تستفيد هذه الطرق من مشاركة الأنماط الفردانية بين الأفراد على مدى فترات قصيرة من التسلسل لحساب الأليلات. تتضمن حزم البرامج الحالية الخاصة بتضمين النمط الجيني IMPUTE2[19] و Minimac و Beagle و MaCH. [20][21]
بالإضافة إلى حساب الارتباط، من الشائع مراعاة أي متغيرات قد تؤدي إلى إرباك النتائج. الجنس والعمر أمثلة شائعة للمتغيرات المربكة. علاوة على ذلك، من المعروف أيضًا أن العديد من الاختلافات الجينية مرتبطة بالتجمعات السكانية الجغرافية والتاريخية التي نشأت فيها الطفرات لأول مرة.[22] بسبب هذا الارتباط، يجب أن تأخذ الدراسات في الاعتبار الخلفية الجغرافية والعرقية للمشاركين من خلال التحكم فيما يسمى التقسيم الطبقي للسكان. إذا فشلوا في القيام بذلك، يمكن أن تؤدي هذه الدراسات إلى نتائج إيجابية خاطئة. [23]
بعد حساب نسب الأرجحية وقيم P لجميع أشكال النيوكلوتايد، فإن الطريقة الشائعة هي إنشاء مخطط مانهاتن. في سياق دراسات الترابط الجينومي الكامل، تُظهِر هذه المؤامرة اللوغاريتم السلبي للقيمة P كدالّة للموقع الجيني. وهكذا يبرُز النوكليوتيدات المفردة متعددة الأشكال مع الارتباط الأكثر أهمية في الحبكة، وعادةً ما تكون بمثابة أكوام من النقاط بسبب بنية الهابلوبلوك (Haploblock structure). الأهم من ذلك، تُصحَّح بداية القيمة P للأهمية لمشاكل الاختبار المتعددة. تختلف العتبة الدقيقة حسب الدراسة،[24] ولكن الحد التقليدي هو 5×10−8 ليكون مهمًا في مواجهة مئات الآلاف إلى الملايين من تعدد أشكال النوكليوتيدات المفردة المُختبَرة.[11][12][25] تقوم دراسات الترابط الجينومي الكامل عادةً بإجراء التحليل الأول في مجموعة اكتشاف، متبوعًا بالتحقق من صحة أهم النوكليوتيدات المفردة متعددة الأشكال في مجموعة تحقق مستقلة.
النتائج
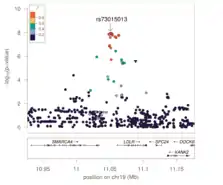
بُذِلت محاولات لإنشاء كتالوجات شاملة للنوكليوتيدات المفردة متعددة الأشكال التي حُدِّدَت من دراسات الترابط الجينومي الكامل.[27] اعتبارًا من عام 2009، بلغ تعداد النيوكلوتايد المرتبط بالأمراض بالآلاف. [28]
قارنت دراسة الترابط الجينومي الكامل الأولى، التي أُجريَت في عام 2005، 96 مريضًا يعانون من التنكس البقعي المرتبط بالعمر مع 50 عنصر تحكم صحي،[29] فحدَّدت اثنين من النوكليوتيدات المفردة متعددة الأشكال مع تردد أليل متغير بشكل كبير بين المجموعتين. تم العثور على أشكال النوكليوتيدات هذه في عامل مُكمِّل تشفير الجينات H، والذي كان اكتشافًا غير متوقع في بحث التنكس البقعي. وقد دفعت نتائج دراسات الترابط الجينومي الكامل الأولى في وقت لاحق إلى مزيد من البحث الوظيفي نحو التلاعب العلاجي للنظام التكميلي في التنكس البقعي.[30] منشور بارز آخر في تاريخ دراسات الترابط الجينومي الكامل هو دراسة Wellcome Trust Case Control Consortium (WTCCC)، وهي أكبر دراسة ترابط جينومي كامل أُجريَت على الإطلاق في وقت نشرها في عام 2007. تضمنت هذه الدراسة 14000 حالة من سبعة أمراض شائعة (حوالي 2000 فرد لكلٍ من مرض القلب التاجي والسكري من النوع 1 والسكري من النوع 2 والتهاب المفاصل الروماتويدي ومرض كرون والاضطراب ثنائي القطب وارتفاع ضغط الدم) و 3000 عنصر تحكم مشترك. نجحت هذه الدراسة في الكشف عن العديد من الجينات المرضية الجديدة الكامنة وراء هذه الأمراض. [31][13]
منذ هذه الدراسات التاريخية الأولى للترابط الجينومي الكامل، كان هناك اتجاهان عامان.[32] كان أحدها نحو أحجام عينات أكبر وأكبر. في عام 2018، وصلت العديد من دراسات الارتباط على مستوى الجينوم إلى حجم عينة إجمالي يزيد عن مليون مشارك، بما في ذلك 1.1 مليون في دراسة على مستوى الجينوم للتحصيل التعليمي[33] ودراسة للأرق تحتوي على 1.3 مليون فرد.[34] والسبب هو الدافع نحو الكشف الموثوق به عن تعدد أشكال النيوكلوتايد للمخاطر التي لها نسب أرجحية أقل وتردد أليل أقل. كان هناك اتجاه آخر نحو استخدام أنماط ظاهرية محددة بدقة، مثل دهون الدم، والبرونسولين أو المؤشرات الحيوية المماثلة.[35][36] وتسمى هذه الأنماط الظاهرية الوسيطة، وقد تكون تحليلاتها ذات قيمة للبحث الوظيفي في المؤشرات الحيوية.[37] تستخدم مجموعة متنوعة من دراسات الترابط الجينومي الكامل مشاركين هم أقارب من الدرجة الأولى للأشخاص المصابين بمرض، سُمِّيَ هذا النوع من الدراسة بدراسة الارتباط على مستوى الجينوم بالوكالة (GWAX). [38]
كانت النقطة المركزية للنقاش حول دراسات الترابط الجينومي الكامل هي أن معظم الاختلافات في أشكال النوكليوتيدات المفردة المتعددة التي وجدتها دراسات الترابط الجينومي الكامل مرتبطة فقط بزيادة خطر الإصابة بالمرض، وليس لها سوى قيمة تنبؤية صغيرة. متوسط نسبة الأرجحية هو 1.33 لكل مخاطرة نوكليوتيدات المفردة متعددة الأشكال، مع وجود عدد قليل فقط يُظهِر نسب الأرجحية أعلى من 3.0.[39][40] تُعتبر هذه المقادير صغيرة لأنها لا تفسر الكثير من التباين الوراثي. قُدِّر هذا الاختلاف الموروث من دراسات التوريث على أساس التوائم أحادية الزيجوت. على سبيل المثال، من المعروف أن 80-90٪ من التباين في الطول يمكن تفسيره بالاختلافات الوراثية، لكن دراسات الترابط الجينومي الكامل لا تمثل سوى أقلية من هذا التباين. [41]
التطبيقات السريرية
يتمثل التحدي الذي يواجه دراسة الترابط الجينومي الكامل الناجحة في المستقبل في تطبيق النتائج بطريقة تسرع تطوير الأدوية والتشخيص، بما في ذلك دمج أفضل للدراسات الجينية في عملية تطوير الدواء والتركيز على دور التباين الجيني في الحفاظ على الصحة كمخطط أساسي لتصميم أدوية وتشخيصات جديدة.[42] لقد نظرت العديد من الدراسات في استخدام علامات المُخاطَرة النوكليوتيدات المفردة متعددة الأشكال كوسيلة لتحسين دقة التشخيص بشكل مباشر. وجد البعض أن دقة التشخيص تتحسن،[43] بينما أفاد آخرون بفوائد طفيفة فقط من هذا الاستخدام.[44] بشكل عام، تتمثل مشكلة هذا النهج المباشر في المقادير الصغيرة للتأثيرات المُلاحَظة. التأثير الصغير يُترجَم في النهاية إلى فصل ضعيف للحالات والضوابط، وبالتالي فقط تحسن بسيط في دقة التشخيص. ولذلك فإن التطبيق البديل هو إمكانية دراسات الترابط الجينومي الكامل لتوضيح الفزيولوجيا المرضية. [45]
يرتبط أحد هذه النجاحات بتحديد المتغير الجيني المرتبط بالاستجابة للعلاج المضاد لفيروس التهاب الكبد سي. بالنسبة إلى النمط الجيني 1 من التهاب الكبد C المُعالج بـ بيغنتيرفيرون – ألفا – 2أ أو بيغنتيرفيرون – ألفا – 2ب جنبًا إلى جنب مع الريبافيرين، أظهرت دراسة الترابط الجينومي الكامل أن النوكليوتيدات المفردة متعددة الأشكال بالقرب من جين IL28B البشري، الذي يشفر الإنترفيرون لامدا 3 (Interferon lambda 3)، يرتبط باختلافات كبيرة استجابة للعلاج. أظهر تقرير لاحق أن نفس المتغيرات الجينية مرتبطة أيضًا بالتخلص الطبيعي من النمط الجيني 1 لفيروس التهاب الكبد سي. سهلت هذه النتائج الرئيسية تطوير الطب الشخصي وسمحت للأطباء بتخصيص القرارات الطبية بناءً على النمط الجيني للمريض. [46][47][48]
لقد أدى هدف توضيح الفيزيولوجيا المرضية أيضًا إلى زيادة الاهتمام بالارتباط بين مخاطر النيوكلوتايد والتعبير الجيني للجينات القريبة، ما يُسمَّى بدراسات مواضع السمات الكمية للتعبير (Expression quantitative trait loci).[49] والسبب هو أن دراسات الترابط الجينومي الكامل تحدد مخاطر النيوكلوتايد، ولكن ليس الجينات الخطرة، وأن مواصفات الجينات هي خطوة واحدة نحو أهداف دوائية قابلة للتنفيذ. نتيجة لذلك، تضمنت دراسات الترابط الجينومي الكامل الرئيسية بحلول عام 2011 تحليلاً موسعًا لمواضع السمات الكمية للتعبير.[50][51][52] يعد موضع سورتلين 1 (Sortilin 1) أحد أقوى تأثيرات مواضع السمات الكمية للتعبير التي لوحظت بالنسبة لمخاطر النوكليوتيدات المفردة متعددة الأشكال التي حُدِّدَت بواسطة دراسة الترابط الجينومي الكامل. وقد سلَّطت دراسات المتابعة الوظيفية لهذا الموضع باستخدام أحماض نووية ريبوزية متداخلة صغيرة (RNA) وفئران معطلة الجينات الضوء على عملية التمثيل الغذائي للبروتينات الدهنية منخفضة الكثافة، والتي لها آثار سريرية مهمة على أمراض القلب الوعائية. [36][53][54]
الرجفان الأذيني
على سبيل المثال، كشف التحليل التلوي الذي أُجرِيَ في عام 2018 عن اكتشاف 70 موقعًا جديدًا مرتبطًا بالرجفان الأذيني. حُدِّدَت المتغيرات المختلفة المرتبطة بجينات ترميز عامل النسخ، مثل TBX3 و TBX5، والتي تشارك في تنظيم التوصيل القلبي، في تعديل القناة الأيونية وتطور القلب. كما حُدِّدَت جينات جديدة تشارك في تسرع القلب أو مرتبطة بتغيير اتصالات الخلايا العضلية القلبية. [55]
التطبيقات الزراعية
مراحل نمو النبات ومكونات المحصول
تعمل دراسات الترابط الجينومي الكامل كأداة مهمة في تربية النبات. مع بيانات التنميط الجيني والأنماط الظاهرية الكبيرة، تعد دراسة الترابط الجينومي الكامل قوية في تحليل أنماط الوراثة المعقدة للسمات التي تعد مكونات محصول مهمة مثل عدد الحبوب في كل سنبلة ووزن كل حبة وهيكل نبات. في دراسة ترابط جينومي كامل على القمح الربيعي، كشفت هذه الدراسة عن وجود علاقة قوية بين إنتاج الحبوب وبيانات التمهيد والكتلة الحيوية وعدد الحبوب لكل سنبلة. [56]
مسببات الأمراض النباتية
شكّل ظهور مسببات الأمراض النباتية تهديدات خطيرة لصحة النبات والتنوع البيولوجي. تحت هذا الاعتبار، قد يكون تحديد الأنواع البرية التي لديها مقاومة طبيعية لبعض مسببات الأمراض ذات أهمية حيوية. علاوة على ذلك، نحن بحاجة للتنبؤ بأي الأليلات مرتبطة بالمقاومة. تعد دراسات الترابط الجينومي الكامل أداة قوية لاكتشاف العلاقات بين متغيرات معينة ومقاومة مسببات الأمراض النباتية، وهو أمر مفيد لتطوير أصناف جديدة مقاومة للعوامل الممرضة. [57]
هوامش
- ملاحظة 1 من الإنجليزية Genome-wide association study
مراجع
- "Genomewide association studies and assessment of the risk of disease". The New England Journal of Medicine. 363 (2): 166–76. July 2010. doi:10.1056/NEJMra0905980. PMID 20647212. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "How to interpret a genome-wide association study". JAMA. 299 (11): 1335–44. March 2008. doi:10.1001/jama.299.11.1335. PMID 18349094. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Genome-Wide Association Studies". National Human Genome Research Institute. مؤرشف من الأصل في 1 فبراير 2019. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Human molecular genetics (الطبعة 4th ed). New York: Garland Science. 2011. ISBN 978-0-8153-4149-9. OCLC 477270509. مؤرشف من الأصل في 20 يوليو 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة)صيانة CS1: نص إضافي (link) - "OMIM - Online Mendelian Inheritance in Man". web.archive.org. 2011-12-05. اطلع عليه بتاريخ 06 فبراير 2021. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Altmüller, J.; Palmer, L. J.; Fischer, G.; Scherb, H.; Wjst, M. (2001-11). "Genomewide scans of complex human diseases: true linkage is hard to find". American Journal of Human Genetics. 69 (5): 936–950. doi:10.1086/324069. ISSN 0002-9297. PMID 11565063. مؤرشف من الأصل في 20 أغسطس 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Risch, N.; Merikangas, K. (1996-09-13). "The future of genetic studies of complex human diseases". Science (New York, N.Y.). 273 (5281): 1516–1517. doi:10.1126/science.273.5281.1516. ISSN 0036-8075. PMID 8801636. مؤرشف من الأصل في 23 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Greely, Henry T. (2007). "The uneasy ethical and legal underpinnings of large-scale genomic biobanks". Annual Review of Genomics and Human Genetics. 8: 343–364. doi:10.1146/annurev.genom.7.080505.115721. ISSN 1527-8204. PMID 17550341. مؤرشف من الأصل في 28 أغسطس 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - International HapMap Consortium (2003-12-18). "The International HapMap Project". Nature. 426 (6968): 789–796. doi:10.1038/nature02168. ISSN 1476-4687. PMID 14685227. مؤرشف من الأصل في 16 أكتوبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Schena, M.; Shalon, D.; Davis, R. W.; Brown, P. O. (1995-10-20). "Quantitative monitoring of gene expression patterns with a complementary DNA microarray". Science (New York, N.Y.). 270 (5235): 467–470. doi:10.1126/science.270.5235.467. ISSN 0036-8075. PMID 7569999. مؤرشف من الأصل في 23 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Bush, William S.; Moore, Jason H. (2012). "Chapter 11: Genome-wide association studies". PLoS computational biology. 8 (12): e1002822. doi:10.1371/journal.pcbi.1002822. ISSN 1553-7358. PMID 23300413. مؤرشف من الأصل في 04 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Clarke, Geraldine M.; Anderson, Carl A.; Pettersson, Fredrik H.; Cardon, Lon R.; Morris, Andrew P.; Zondervan, Krina T. (2011-02). "Basic statistical analysis in genetic case-control studies". Nature Protocols. 6 (2): 121–133. doi:10.1038/nprot.2010.182. ISSN 1750-2799. PMID 21293453. مؤرشف من الأصل في 17 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Wellcome Trust Case Control Consortium (2007-06-07). "Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls". Nature. 447 (7145): 661–678. doi:10.1038/nature05911. ISSN 1476-4687. PMID 17554300. مؤرشف من الأصل في 29 أكتوبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Purcell, Shaun; Neale, Benjamin; Todd-Brown, Kathe; Thomas, Lori; Ferreira, Manuel A. R.; Bender, David; Maller, Julian; Sklar, Pamela; de Bakker, Paul I. W. (2007-09). "PLINK: a tool set for whole-genome association and population-based linkage analyses". American Journal of Human Genetics. 81 (3): 559–575. doi:10.1086/519795. ISSN 0002-9297. PMID 17701901. مؤرشف من الأصل في 17 يناير 2021. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Llinares-López, Felipe; Grimm, Dominik G.; Bodenham, Dean A.; Gieraths, Udo; Sugiyama, Mahito; Rowan, Beth; Borgwardt, Karsten (2015-06-15). "Genome-wide detection of intervals of genetic heterogeneity associated with complex traits". Bioinformatics (Oxford, England). 31 (12): i240–249. doi:10.1093/bioinformatics/btv263. ISSN 1367-4811. PMID 26072488. مؤرشف من الأصل في 30 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Ayati, Marzieh; Erten, Sinan; Chance, Mark R.; Koyutürk, Mehmet (2015-12). "MOBAS: identification of disease-associated protein subnetworks using modularity-based scoring". EURASIP journal on bioinformatics & systems biology. 2015: 7. doi:10.1186/s13637-015-0025-6. ISSN 1687-4145. PMID 28194175. مؤرشف من الأصل في 07 أكتوبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Ayati, Marzieh; Koyutürk, Mehmet (2015-09-09). "Assessing the collective disease association of multiple genomic loci". Proceedings of the 6th ACM Conference on Bioinformatics, Computational Biology and Health Informatics. Atlanta, Georgia: Association for Computing Machinery: 376–385. doi:10.1145/2808719.2808758. ISBN 978-1-4503-3853-0. مؤرشف من الأصل في 3 فبراير 2021. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Marchini, Jonathan; Howie, Bryan (2010-07). "Genotype imputation for genome-wide association studies". Nature Reviews. Genetics. 11 (7): 499–511. doi:10.1038/nrg2796. ISSN 1471-0064. PMID 20517342. مؤرشف من الأصل في 15 ديسمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Howie, Bryan; Marchini, Jonathan; Stephens, Matthew (2011-11). "Genotype imputation with thousands of genomes". G3 (Bethesda, Md.). 1 (6): 457–470. doi:10.1534/g3.111.001198. ISSN 2160-1836. PMID 22384356. مؤرشف من الأصل في 04 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Browning, Brian L.; Browning, Sharon R. (2009-02). "A unified approach to genotype imputation and haplotype-phase inference for large data sets of trios and unrelated individuals". American Journal of Human Genetics. 84 (2): 210–223. doi:10.1016/j.ajhg.2009.01.005. ISSN 1537-6605. PMID 19200528. مؤرشف من الأصل في 15 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Li, Yun; Willer, Cristen J.; Ding, Jun; Scheet, Paul; Abecasis, Gonçalo R. (2010-12). "MaCH: using sequence and genotype data to estimate haplotypes and unobserved genotypes". Genetic Epidemiology. 34 (8): 816–834. doi:10.1002/gepi.20533. ISSN 1098-2272. PMID 21058334. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Novembre, John; Johnson, Toby; Bryc, Katarzyna; Kutalik, Zoltán; Boyko, Adam R.; Auton, Adam; Indap, Amit; King, Karen S.; Bergmann, Sven (2008-11-06). "Genes mirror geography within Europe". Nature. 456 (7218): 98–101. doi:10.1038/nature07331. ISSN 1476-4687. PMID 18758442. مؤرشف من الأصل في 15 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Charney, Evan (2017-01). "Genes, behavior, and behavior genetics". Wiley Interdisciplinary Reviews. Cognitive Science. 8 (1–2). doi:10.1002/wcs.1405. ISSN 1939-5086. PMID 27906529. مؤرشف من الأصل في 06 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Wittkowski, K. M.; Sonakya, V.; Bigio, B.; Tonn, M. K.; Shic, F.; Ascano, M.; Nasca, C.; Gold-Von Simson, G. (2014-01-28). "A novel computational biostatistics approach implies impaired dephosphorylation of growth factor receptors as associated with severity of autism". Translational Psychiatry. 4: e354. doi:10.1038/tp.2013.124. ISSN 2158-3188. PMID 24473445. مؤرشف من الأصل في 05 أكتوبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Barsh, Gregory S.; Copenhaver, Gregory P.; Gibson, Greg; Williams, Scott M. (2012-07). "Guidelines for genome-wide association studies". PLoS genetics. 8 (7): e1002812. doi:10.1371/journal.pgen.1002812. ISSN 1553-7404. PMID 22792080. مؤرشف من الأصل في 26 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Sanna, Serena; Li, Bingshan; Mulas, Antonella; Sidore, Carlo; Kang, Hyun M.; Jackson, Anne U.; Piras, Maria Grazia; Usala, Gianluca; Maninchedda, Giuseppe (2011-07). "Fine mapping of five loci associated with low-density lipoprotein cholesterol detects variants that double the explained heritability". PLoS genetics. 7 (7): e1002198. doi:10.1371/journal.pgen.1002198. ISSN 1553-7404. PMID 21829380. مؤرشف من الأصل في 04 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Hindorff, Lucia A.; Sethupathy, Praveen; Junkins, Heather A.; Ramos, Erin M.; Mehta, Jayashri P.; Collins, Francis S.; Manolio, Teri A. (2009-06-09). "Potential etiologic and functional implications of genome-wide association loci for human diseases and traits". Proceedings of the National Academy of Sciences of the United States of America. 106 (23): 9362–9367. doi:10.1073/pnas.0903103106. ISSN 1091-6490. PMID 19474294. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Johnson, Andrew D.; O'Donnell, Christopher J. (2009-01-22). "An open access database of genome-wide association results". BMC medical genetics. 10: 6. doi:10.1186/1471-2350-10-6. ISSN 1471-2350. PMID 19161620. مؤرشف من الأصل في 15 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Haines, Jonathan L.; Hauser, Michael A.; Schmidt, Silke; Scott, William K.; Olson, Lana M.; Gallins, Paul; Spencer, Kylee L.; Kwan, Shu Ying; Noureddine, Maher (2005-04-15). "Complement factor H variant increases the risk of age-related macular degeneration". Science (New York, N.Y.). 308 (5720): 419–421. doi:10.1126/science.1110359. ISSN 1095-9203. PMID 15761120. مؤرشف من الأصل في 29 أكتوبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Fridkis-Hareli, Masha; Storek, Michael; Mazsaroff, Istvan; Risitano, Antonio M.; Lundberg, Ante S.; Horvath, Christopher J.; Holers, V. Michael (2011-10-27). "Design and development of TT30, a novel C3d-targeted C3/C5 convertase inhibitor for treatment of human complement alternative pathway-mediated diseases". Blood. 118 (17): 4705–4713. doi:10.1182/blood-2011-06-359646. ISSN 1528-0020. PMID 21860027. مؤرشف من الأصل في 04 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Press Release - 6th June 2007 - WTCCC". www.wtccc.org.uk. مؤرشف من الأصل في 04 أغسطس 2020. اطلع عليه بتاريخ 06 فبراير 2021. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Ioannidis, John P. A.; Thomas, Gilles; Daly, Mark J. (2009-05). "Validating, augmenting and refining genome-wide association signals". Nature Reviews. Genetics. 10 (5): 318–329. doi:10.1038/nrg2544. ISSN 1471-0064. PMID 19373277. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Lee, James J.; Wedow, Robbee; Okbay, Aysu; Kong, Edward; Maghzian, Omeed; Zacher, Meghan; Nguyen-Viet, Tuan Anh; Bowers, Peter; Sidorenko, Julia (07 23, 2018). "Gene discovery and polygenic prediction from a genome-wide association study of educational attainment in 1.1 million individuals". Nature Genetics. 50 (8): 1112–1121. doi:10.1038/s41588-018-0147-3. ISSN 1546-1718. PMID 30038396. مؤرشف من الأصل في 11 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Jansen, Philip R.; Watanabe, Kyoko; Stringer, Sven; Skene, Nathan; Bryois, Julien; Hammerschlag, Anke R.; Leeuw, Christiaan A. de; Benjamins, Jeroen; Muñoz-Manchado, Ana B. (2018-02-01). "Genome-wide Analysis of Insomnia (N=1,331,010) Identifies Novel Loci and Functional Pathways". bioRxiv (باللغة الإنجليزية): 214973. doi:10.1101/214973. مؤرشف من الأصل في 07 يناير 2021. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Strawbridge, Rona J.; Dupuis, Josée; Prokopenko, Inga; Barker, Adam; Ahlqvist, Emma; Rybin, Denis; Petrie, John R.; Travers, Mary E.; Bouatia-Naji, Nabila (2011-10). "Genome-wide association identifies nine common variants associated with fasting proinsulin levels and provides new insights into the pathophysiology of type 2 diabetes". Diabetes. 60 (10): 2624–2634. doi:10.2337/db11-0415. ISSN 1939-327X. PMID 21873549. مؤرشف من الأصل في 27 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Kathiresan, Sekar; Willer, Cristen J.; Peloso, Gina M.; Demissie, Serkalem; Musunuru, Kiran; Schadt, Eric E.; Kaplan, Lee; Bennett, Derrick; Li, Yun (2009-01). "Common variants at 30 loci contribute to polygenic dyslipidemia". Nature Genetics. 41 (1): 56–65. doi:10.1038/ng.291. ISSN 1546-1718. PMID 19060906. مؤرشف من الأصل في 15 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Danesh, John; Pepys, Mark B. (2009-11-24). "C-reactive protein and coronary disease: is there a causal link?". Circulation. 120 (21): 2036–2039. doi:10.1161/CIRCULATIONAHA.109.907212. ISSN 1524-4539. PMID 19901186. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Liu, Jimmy Z.; Erlich, Yaniv; Pickrell, Joseph K. (2017-03). "Case-control association mapping by proxy using family history of disease". Nature Genetics. 49 (3): 325–331. doi:10.1038/ng.3766. ISSN 1546-1718. PMID 28092683. مؤرشف من الأصل في 16 أكتوبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Ku, Chee Seng; Loy, En Yun; Pawitan, Yudi; Chia, Kee Seng (2010-04). "The pursuit of genome-wide association studies: where are we now?". Journal of Human Genetics. 55 (4): 195–206. doi:10.1038/jhg.2010.19. ISSN 1435-232X. PMID 20300123. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Manolio, Teri A. (2010-07-08). "Genomewide association studies and assessment of the risk of disease". The New England Journal of Medicine. 363 (2): 166–176. doi:10.1056/NEJMra0905980. ISSN 1533-4406. PMID 20647212. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Maher, Brendan (2008-11-06). "Personal genomes: The case of the missing heritability". Nature. 456 (7218): 18–21. doi:10.1038/456018a. ISSN 1476-4687. PMID 18987709. مؤرشف من الأصل في 15 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Iadonato, Shawn P.; Katze, Michael G. (2009-09-17). "Genomics: Hepatitis C virus gets personal". Nature. 461 (7262): 357–358. doi:10.1038/461357a. ISSN 1476-4687. PMID 19759611. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Muehlschlegel, Jochen D.; Liu, Kuang-Yu; Perry, Tjörvi E.; Fox, Amanda A.; Collard, Charles D.; Shernan, Stanton K.; Body, Simon C.; CABG Genomics Investigators (2010-09-14). "Chromosome 9p21 variant predicts mortality after coronary artery bypass graft surgery". Circulation. 122 (11 Suppl): S60–65. doi:10.1161/CIRCULATIONAHA.109.924233. ISSN 1524-4539. PMID 20837927. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Paynter, Nina P.; Chasman, Daniel I.; Paré, Guillaume; Buring, Julie E.; Cook, Nancy R.; Miletich, Joseph P.; Ridker, Paul M. (2010-02-17). "Association between a literature-based genetic risk score and cardiovascular events in women". JAMA. 303 (7): 631–637. doi:10.1001/jama.2010.119. ISSN 1538-3598. PMID 20159871. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Couzin-Frankel, Jennifer (2010-06-04). "Major heart disease genes prove elusive". Science (New York, N.Y.). 328 (5983): 1220–1221. doi:10.1126/science.328.5983.1220. ISSN 1095-9203. PMID 20522751. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Ge, Dongliang; Fellay, Jacques; Thompson, Alexander J.; Simon, Jason S.; Shianna, Kevin V.; Urban, Thomas J.; Heinzen, Erin L.; Qiu, Ping; Bertelsen, Arthur H. (2009-09-17). "Genetic variation in IL28B predicts hepatitis C treatment-induced viral clearance". Nature. 461 (7262): 399–401. doi:10.1038/nature08309. ISSN 1476-4687. PMID 19684573. مؤرشف من الأصل في 26 يناير 2021. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Thomas, David L.; Thio, Chloe L.; Martin, Maureen P.; Qi, Ying; Ge, Dongliang; O'Huigin, Colm; Kidd, Judith; Kidd, Kenneth; Khakoo, Salim I. (2009-10-08). "Genetic variation in IL28B and spontaneous clearance of hepatitis C virus". Nature. 461 (7265): 798–801. doi:10.1038/nature08463. ISSN 1476-4687. PMID 19759533. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Lu, Yi-Fan; Goldstein, David B.; Angrist, Misha; Cavalleri, Gianpiero (2014-07-24). "Personalized medicine and human genetic diversity". Cold Spring Harbor Perspectives in Medicine. 4 (9): a008581. doi:10.1101/cshperspect.a008581. ISSN 2157-1422. PMID 25059740. مؤرشف من الأصل في 18 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Folkersen, Lasse; van't Hooft, Ferdinand; Chernogubova, Ekaterina; Agardh, Hanna E.; Hansson, Göran K.; Hedin, Ulf; Liska, Jan; Syvänen, Ann-Christine; Paulsson-Berne, Gabrielle (2010-08). "Association of genetic risk variants with expression of proximal genes identifies novel susceptibility genes for cardiovascular disease". Circulation. Cardiovascular Genetics. 3 (4): 365–373. doi:10.1161/CIRCGENETICS.110.948935. ISSN 1942-3268. PMID 20562444. مؤرشف من الأصل في 16 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Bown, Matthew J.; Jones, Gregory T.; Harrison, Seamus C.; Wright, Benjamin J.; Bumpstead, Suzannah; Baas, Annette F.; Gretarsdottir, Solveig; Badger, Stephen A.; Bradley, Declan T. (2011-11-11). "Abdominal aortic aneurysm is associated with a variant in low-density lipoprotein receptor-related protein 1". American Journal of Human Genetics. 89 (5): 619–627. doi:10.1016/j.ajhg.2011.10.002. ISSN 1537-6605. PMID 22055160. مؤرشف من الأصل في 04 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Coronary Artery Disease (C4D) Genetics Consortium (2011-03-06). "A genome-wide association study in Europeans and South Asians identifies five new loci for coronary artery disease". Nature Genetics. 43 (4): 339–344. doi:10.1038/ng.782. ISSN 1546-1718. PMID 21378988. مؤرشف من الأصل في 17 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Johnson, Toby; Gaunt, Tom R.; Newhouse, Stephen J.; Padmanabhan, Sandosh; Tomaszewski, Maciej; Kumari, Meena; Morris, Richard W.; Tzoulaki, Ioanna; O'Brien, Eoin T. (2011-12-09). "Blood pressure loci identified with a gene-centric array". American Journal of Human Genetics. 89 (6): 688–700. doi:10.1016/j.ajhg.2011.10.013. ISSN 1537-6605. PMID 22100073. مؤرشف من الأصل في 04 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Dubé, Joseph B.; Johansen, Christopher T.; Hegele, Robert A. (2011-06). "Sortilin: an unusual suspect in cholesterol metabolism: from GWAS identification to in vivo biochemical analyses, sortilin has been identified as a novel mediator of human lipoprotein metabolism". BioEssays: News and Reviews in Molecular, Cellular and Developmental Biology. 33 (6): 430–437. doi:10.1002/bies.201100003. ISSN 1521-1878. PMID 21462369. مؤرشف من الأصل في 17 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Bauer, Robert C.; Stylianou, Ioannis M.; Rader, Daniel J. (2011-04). "Functional validation of new pathways in lipoprotein metabolism identified by human genetics". Current Opinion in Lipidology. 22 (2): 123–128. doi:10.1097/MOL.0b013e32834469b3. ISSN 1473-6535. PMID 21311327. مؤرشف من الأصل في 17 نوفمبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Roselli, Carolina; Chaffin, Mark D.; Weng, Lu-Chen; Aeschbacher, Stefanie; Ahlberg, Gustav; Albert, Christine M.; Almgren, Peter; Alonso, Alvaro; Anderson, Christopher D. (06 11, 2018). "Multi-ethnic genome-wide association study for atrial fibrillation". Nature Genetics. 50 (9): 1225–1233. doi:10.1038/s41588-018-0133-9. ISSN 1546-1718. PMID 29892015. مؤرشف من الأصل في 20 أكتوبر 2020. الوسيط
|CitationClass=تم تجاهله (مساعدة); تحقق من التاريخ في:|تاريخ=(مساعدة) - Turuspekov, Yerlan; Baibulatova, Aida; Yermekbayev, Kanat; Tokhetova, Laura; Chudinov, Vladimir; Sereda, Grigoriy; Ganal, Martin; Griffiths, Simon; Abugalieva, Saule (2017-11-14). "GWAS for plant growth stages and yield components in spring wheat (Triticum aestivum L.) harvested in three regions of Kazakhstan". BMC plant biology. 17 (Suppl 1): 190. doi:10.1186/s12870-017-1131-2. ISSN 1471-2229. PMID 29143598. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Bartoli, Claudia; Roux, Fabrice (2017). "Genome-Wide Association Studies In Plant Pathosystems: Toward an Ecological Genomics Approach". Frontiers in Plant Science. 8: 763. doi:10.3389/fpls.2017.00763. ISSN 1664-462X. PMID 28588588. الوسيط
|CitationClass=تم تجاهله (مساعدة)
- بوابة طب
- بوابة علم الأحياء
 صور وملفات صوتية من كومنز
صور وملفات صوتية من كومنز