تماثل متسلسل
التماثل المتسلسل هو التماثل البيولوجي بين تسلسل الدنا أو الرنا أو سلاسل البروتين، المعرّفة من حيث النسب المشترك في تاريخ الحياة التطوّري. يمكن ان يتشارك قسمان من الحمض النووي أسلافهما بسبب ثلاث ظواهر: حدث انتواعيّ أو حدث تكراريّ أو حدث نقل الجينات الأفقيّ.[1]
عادةً ما يتم استنتاج التماثل بين الدنا والرنا والبروتينات من التشابه في تسلسل النوكليوتيدات أو الأحماض الأمينيّة. التشابه الكبير هو دليل قويّ على أن التسلسلين مرتبطان بالتغيّرات التطوريّة من تسلسل الأسلاف المشتركة. يتم استخدام محاذاة تسلسلات متعدّدة من أجل الإشارة إلى أي منطقة متجانسة من كل تسلسل.
الهويّة والتشابه والحفظ
غالباً ما يستخدم مصطلح "التماثل المئوي" ليعني "تشابه السلسلة". النسبة المئويّة للمخلّفات المتطابقة (النسبة المئويّة للهويّة) أو النسبة المئويّة للمخلّفات المحفوظة بخصائص فيزيائيّة وكيميائيّة متماثلة (تشابه النسبة المئويّة)، على سبيل المثال "ليوسين" و"إيزوليوسين"، وعادةً ما يستخدم "لتحديد التماثل". استناداً إلى تعريف التماثل المحدّد أعلاه يعتبر هذا المصطلح غير صحيح لأن تشابه التسلسل هو الملاحظة، أما التماثل فهو الاستنتاج. التسلسلات إمّا أن تكون متجانسة أو لا.
كما هو الحال مع الهياكل المورفوجيولوجيّة والتشريحيّة، قد يحدث تشابه التسلسل بسبب تطوّر متقارب، أو كما في التسلسلات الأقصر، عن طريق الصدفة، مما يعني أنها بالأصل ليست متجانسة. وتسمّى أيضاً السلاسل المتشابهة بالسلاسل المحفوظة. لا يجب الخلط بين ما سبق وبين الحفظ في سلاسل الأحماض الأمينيّة، حيث يتمّ استبدال الحمض الأميني في موضع معيّن بحمض مختلف له خصائص فيزيائيّة وكيميائيّة مكافئة وظيفيّاً.
يمكن أن يحدث التماثل الجزئي عندما يكون لجزء من التتابعات المقارنة أصل مشترك، بينما الباقي ليسوا كذلك. قد ينتج هذا التماثل الجزئي عن حدث اندماج الجينات.
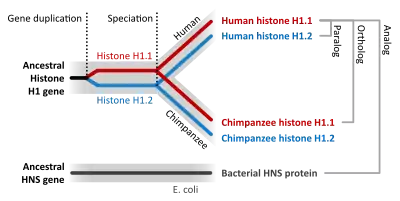
أورثولوجي
في الأعلى: يتكرّر جين من الأسلاف لإنتاج اثنين من نظرائه (الجينات A وB). ينتج عن حدوث تباين أورثولوجي في النوعين الناتجين. في الأسفل: في الأنواع المنفصلة، يكون للجين غير ذي الصلة وظيفة مماثلة (الجين C) ولكن له أصل تطوّري منفصل وكذلك متناظر.
تكون السلاسل المتماثلة متجانسة إذا تمّ استنتاج أنّها تنحدر من نفس تسلسل الأجداد ولكن محدّدة بحدث انتواعيّ: عندما يتحوّل نوع ما إلى نوعين منفصلين، تعتبر نسخ الجين الواحد في النوعين الناتجين جينات أورثولوجيّة. الجينات الأورثولوجيّة، هي جينات في أنواع مختلفة نشأت عن الأصل الأورثولوجي من جين واحد لآخر سلف مشترك. تمّت صياغة مصطلح "أورثولوجيّ" في عام 1970 من قبل عالم التطوّر الجزيئي والتر فيتش.[2]
على سبيل المثال، يوجد البروتين التنظيميّ للنبات والموجود في نبات الأرابيدوبسيس (النبات عالي الخلايا) والكلاميدوموناز (الطحالب الخضراء ذات الخليّة الواحدة). إصدار الكلاميدوموناز أكثر تعقيداً: فهو يعبر الغشاء مرّتين بدلاً من مرّة واحدة، ويحتوي على مجالات إضافيّة ويخضع لربط بديل. مع ذلك، يمكن أن يحلّ محلّ بروتين أرابيدوبسيس بشكل أبسط بكثير، إذا تمّ نقله من الطحالب إلى الجينوم النباتي عن طريق الهندسة الوراثيّة. يشير التشابه المتسلسل الهامّ والمجالات الوظيفيّة المشتركة إلى أن هذين الجينين هما جينات أورثولوجيّة[3]، موروثة من سلف مشترك.
يتمّ تعريف الأورثولوجية بدقّة من حيث النسب. بالنظر إلى أن النسب الدقيق للجينات في الكائنات المختلفة يصعب التأكّد منه بسبب تكرار الجينات وأحداث إعادة ترتيب الجينوم، فإن أقوى دليل على تماثل جينين متشابهين يتم العثور عليه عادةً من خلال إجراء تحليل نسبي للجينات النسبيّة. غالباً ما يكون للأورثولوجيّين نفس الوظيفة.[4]
توفّر التسلسلات المجسّمة معلومات مفيدة في التصنيف ودراسات التطوّر الوراثي للكائنات. يمكن استخدام نمط التباعد الوراثي لتتبع ارتباط الكائنات الحيّة. من المحتمل أن يعرض كيانان مرتبطان ارتباطاً وثيقاً تسلسل الحمض النووي المتماثل جدّاً بين اثنين من الأورثولوجيين. وعلى العكس، فإن الكائن الذي تتمّ إزالته تطوريّاً من كائن حيّ آخر من المحتمل أن يظهر تباعداً أكبر في تسلسل التقويمات التي تتم دراستها.
قواعد بيانات الجينات الأورثولوجيّة
نظراً لأهميّتها الكبيرة في علم الأحياء والمعلوماتيّة الحيوية، فقد تمّ تنظيم الجينات الأورثولوجيّة في العديد من قواعد البيانات المتخصّصة التي توفّر أدوات لتحديد وتحليل تسلسل الجينات الأورثولوجيّة. تستخدم هذه الموارد طرقاً يمكن تصنيفها عموماً في تلك التي تستخدم التحليل الاستدلائي لجميع مقارنات التسلسل الزوجي، وتلك التي تستخدم طرق النشوء والتطوّر. كانت طرق المقارنة التسلسليّة الأولى في قاعدة بيانات سي أو جي إس (COGs) في عام 1997.[5]
النظير المشابه
الجينات المتناظرة، بكل بساطة، هي جينات مرتبطة عبر أحداث التكرار في آخر سلف مشترك (LCA) في الأنواع التي تتم مقارنتها. تنتج عن طفرة الجينات المكرّرة خلال أحداث التكاثر المنفصلة. عندما يشارك المتحدّرون من LCA جينات مكرّرة من الجينات الأصلية بحيث تكون متماثلة ومشتركة، فإن تلك الجينات تعتبر نظيراً مشابهاً.
علم التغاير
التسلسللات المتماثلة الناتجة من النقل الأفقي للجينات تسمى تسلسلات أجنبية متماثلة. يمكن أن تكون للتسلسلات الأجنبية وظائف مختلفة إن كانت البيئة الجديدة مختلفة بشكل كبير عن البيئة السابقة التي كان فيها الجين المنتقل أفقيا. بشكل عام، للتسلسلات المتماثلة الأجنبية وظائف متماثلة في كلا الكائنين. صِيغ هذا المصطلح بواسطة وولتر فيتش.[2]
التكرار
غالباً ما يتم تنظيم النظراء المشابهة بشكل مختلف، على سبيل المثال من خلال وجود أنماط التعبير المختلفة الخاصّة بالأنسجة. ومع ذلك، يمكن أيضاً تنظيمها بشكل مختلف على مستوى البروتين. على سبيل المثال تقوم العصويّة الرقيقة بتشفير اثنين من نظرائها من هيدروجيناز الغلوتامات: يتم نسخ GudB بشكل أساسي بينما يتم تنظيم RocG بإحكام. ومع ذلك، فإن مقايضات الأنزيمات والمروجين تتسبّب في خسائر فادحة، مما يشير إلى تطوّر أنزيم المروّج. يوضّح توصيف البروتينات أن نشاط GudB الأنزيمي، مقارنةً بـRocG، يعتمد بشكل كبير على الغلوتامات ودرجة الحموضة.[6]
مناطق الكروموسومات الشاذة
في بعض الأحيان، تشترك مناطق كبيرة من الكروموسومات في محتوى جيني مماثل لمناطق الكروموسومات الأخرى داخل نفس الجينوم. وتتميّز بشكل جيّد في الجينوم البشري، حيث تمّ استخدامها كدليل لدعم فرضيّة 2R. يتم استنتاج مجموعات من الجينات المكرّرة، والمكرّرة ثلاث مرّات، مع الجينات ذات الصلة مع الكروموسومات المختلفة لتكون بقايا من مضاعفات الجينوم أو الكروموسومات.
المراجع
- Koonin EV (2005). "Orthologs, paralogs, and evolutionary genomics". Annual Review of Genetics. 39: 309–338. doi:10.1146/annurev.genet.39.073003.114725. PMID 16285863. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Fitch WM (June 1970). "Distinguishing homologous from analogous proteins". Systematic Zoology. 19 (2): 99–113. doi:10.2307/2412448. JSTOR 2412448. PMID 5449325. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Fang G, Bhardwaj N, Robilotto R, Gerstein MB (March 2010). "Getting started in gene orthology and functional analysis". PLoS Computational Biology. 6 (3): e1000703. doi:10.1371/journal.pcbi.1000703. PMC 2845645. PMID 20361041. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Falciatore A, Merendino L, Barneche F, Ceol M, Meskauskiene R, Apel K, Rochaix JD (January 2005). "The FLP proteins act as regulators of chlorophyll synthesis in response to light and plastid signals in Chlamydomonas". Genes & Development. 19 (1): 176–187. doi:10.1101/gad.321305. PMC 540235. PMID 15630026. الوسيط
|CitationClass=تم تجاهله (مساعدة) - COGs: Clusters of Orthologous Groups of proteins
Tatusov RL, Koonin EV, Lipman DJ (October 1997). "A genomic perspective on protein families". Science. 278 (5338): 631–637. doi:10.1126/science.278.5338.631. PMID 9381173. مؤرشف من الأصل في 22 مارس 2019. اطلع عليه بتاريخ 12 أبريل 2019. الوسيط|CitationClass=تم تجاهله (مساعدة) - eggNOG: evolutionary genealogy of genes: Non-supervised Orthologous Groups
Muller J, Szklarczyk D, Julien P, Letunic I, Roth A, Kuhn M, Powell S, von Mering C, Doerks T, Jensen LJ, Bork P (January 2010). "eggNOG v2.0: extending the evolutionary genealogy of genes with enhanced non-supervised orthologous groups, species and functional annotations". Nucleic Acids Research. 38 (Database issue): D190–5. doi:10.1093/nar/gkp951. PMC 2808932. PMID 19900971. مؤرشف من الأصل في 17 نوفمبر 2011. اطلع عليه بتاريخ 12 أبريل 2019. الوسيط|CitationClass=تم تجاهله (مساعدة)
- بوابة علم الأحياء الخلوي والجزيئي
- بوابة الكيمياء الحيوية
 صور وملفات صوتية من كومنز
صور وملفات صوتية من كومنز
