ترسيب مناعي للكروماتين
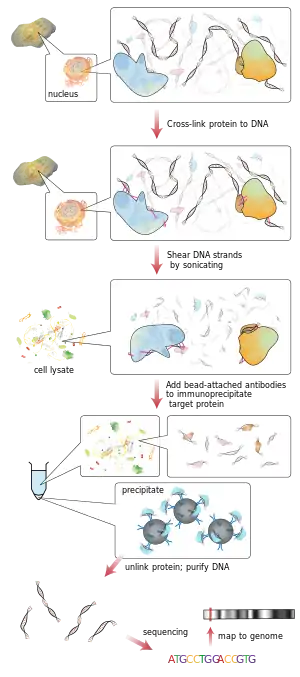
الترسيب المناعي للكروماتين هو تقنية ترسيب مناعي تجريبية تستعمل لتحري التفاعلات بين البروتين و الـ حمض نووي ريبوزي منقوص الأكسجين في الخلية.
يهدف إلى:
- معرفة فيما إذا كانت بروتينات معينة لها علاقة بمناطق معينة على الجينوم مثل علاقة عوامل الانتساخ على المشرع أو غيرها من مناطق الارتباط على الـDNA .
- امكانية تحديد السسترونات.
- تحديد موقع محدد على الجينوم طرأعليه تعديلات بواسطة الهيستون والإشارة إلى الهيستون الهدف الذي قام بالتعديل.[1]
بالمختصر الطريقة كالتالي:
- يُربط البروتين والكروماتين في حلالة الخلية.
- تُجزأ معقد بروتين –DNA (بروتين –كروماتين).
- يتم ترسيب شدف الـDNA مع البروتين/ات المطلوبة منعيا وبشكل نوعي.
- تُنقى شدف الـDNA ويتم تحديد تسلسل النكليوتيدات فيها.
من المفترض ان تكون تسلسل الـDNA هذه مرتبطة مع البروتين الهدف في الخلية.
الترسيب المناعي التقليدي للكروماتين
هناك نوعان أساسيان للترسيب المناعي للكروماتين ChIP يختلفان بشكل أساسي في البدء بتحضير الكروماتين:
- الترسيب المناعي للكروماتين المتصالب (XChIP).
- الترسيب المناعي للكروماتين الفطري (NChIP).
الترسيب المناعي للكروماتين المتصالب (XChIP)
مناسبة لتحديد خريطة DNA عوامل الانتساخ وغيرها من البروتينات المرتبطة بالكروماتين.
- تعتمد هذه الطريقة على استخدام الكروماتين المتصالب القابل للعكس كمادة للبدء حيث يمكن استخدام أشعة الـأشعة فوق البنفسجية[2] أو الفورم الدهيد [3] كعامل لعكس الارتباط المتصالب.
- بعدها يُجزأ الكروماتين المتصالب عادة بالصَوْتَنَة لنحصل على شدف بطول 300- 1000 bp.
- تتم تجزأة الكروماتين باستخدام الفورم الدهيد اللطيف متبوعا بالهضم بواسطة النكلياز إذ أن شدف الكروماتين ذات 400-500 bpتعتبر مناسبة للمقايسة في طريقة الترسيب المناعي للكروماتين كونها تغطي اثنين أو ثلاثة نكليوزومات.[4]
- يثفل حطام الخلايا في الحلالة المجزأة ويتم ترسيب معقد بروتين –DNA مناعيا بشكل انتقائي باستخدام أضاد نوعية للبروتين/ات الهدف وعادة ما يتم ربط هذه الاضداد مع الأغاروز أو السيفاروز أو خرزات مغناطيسية.
- تُجمع معقدات الترسيب المناعي (معقد خرزة- ضد -بروتين- تسلسل الـ DNA الهدف) وتُغسل للتخلص من الكروماتين المرتبط غير النوعي.
- يتم عكس الارتباط المتصالب بين الـDNA والبروتين والتخلص من البروتينات من خلال الهضم بالبروتياز K .
يمكن استخدام المحدد المستضدي الموسوم للبروتين الهدف أو استخدام الاضداد الموسومة بالبيوتين دخل الخلية بدلا من الأضداد للبروتين الفطري الهدف.[5]
- تتم تنقية الـDNA والتعرف عليه بواسطة الـPCR أو microarrays (ChIP-on-chip) أو التنسيل الجزيئي وتحديد تسلسل النكليوتيدات(ChIP-Seq).
الترسيب المناعي للكروماتين الفطري (NChIP)
مناسبة لتحديد موقع الهيستوناتالتي تقوم بالتعديل على الـDNA الهدف.
- عادة يستخدم الكروماتين الفطري ككروماتين للبدء.
- وبما أنو الهيستونات تحيط حول الـDNA كي تشكل النكليوزوم فهي مرتبطة مع الـDNA بشكل طبيعي.
- بعدها يُجزأ الكروماتين بالهضم بواسطة النكلياز الميكروية التي تقطع الـDNA عند طول الرابط تاركة النكليوزومات سليمة ومعطية شدف من الـDNA تحوي من نكليوزوم واحد (200bp) إلى خمسة نكليوزومات (1000bp) بالطول.
- بعدها تُستعمل طرق مشابهة للـXChIP للتخلص من حطام الخلية وترسيب البروتين الهدف وازالة البروتين من معقد الترسيب المناعي وتنقية وتحليل معقد الـDNA المطلوب.
مقارنة بين XChIP وNChIP
الفائدة الأساسية للـNChIPهي في نوعية الضد إذ انه من الضروري ملاحظة أن أغلب أضداد الهيستونات المعدلة متفوقة على المستضدات الصنعية الببتيدية غير الثابتة. كما أن المحدد المستضدي المستعمل في الـXChIP للتمييز قد يتعطل أو يتحطم بارتباط التصالبي مع الفورم ألدهيد. و ان الارتباط التصالبي يتضمن حل المجموعة الامينية على النهايات N مما يسبب تعطيل المحدد المستضدي وهذا يسفر قلة فعالية بروتوكول الـXChIP مقارنة مع الـNChIP. لكن كلا الطريقتين تملك أهدافا وفوائد مختلفة مقارنة ببعضهما فالـXChIP تستعمل لتحديد المواقع الهدف لعوامل الانتساخ وغيرها من البروتينات المرتبطة بالكروماتين بينما الـNChIP تستعمل لتحديد المواقع الهدف للهيستونات المعدلة (انظر الجدول 1).
الجدول 1: فوائد ومساوئXChIP وNChIP
| XChIP | NChIP | |
|---|---|---|
| الفوائد | مناسبة لعوامل الانتساخ أو اي بروتينات ضعيفة الارتباط بالكروماتين .
|
مقياس يستخدم ضد نوعي.
|
| المساوئ | استعادة الكروماتين غير فعالة بسبب تعطل المحدد المستضدي للبروتين الهدف.
|
عادة تكون غير مناسبة للبروتينات غير الهيستونية.
|
تاريخ الـChIP وطرائقها الحديثة
استخدم طالب متخرج في مخبر Lis اشعة أشعة فوق البنفسجية لربط البروتينات تصالبيا وبشكل تساهمي مع الـDNA المجاور في خلايا الجراثيم الحية السليمة. بعد حل الخلايا المتصالبة وترسيب الـRNA بوليمراز الجرثومي مناعيا هجن الـDNA الغني بالـRNA بوليميراز مع مسبابير متناسبة مع اماكن مختلفة من جينات معروفة لتحديد توزع وكثافة البوليميراز عند تلك الجينات في الأحياء. بعد سنة استخدمو الطريقة نفسها لدراسة توزع الـRNA بوليميراز II عند حقيقيات النوى على جينات الصدمة الحرارية في ذبابة الفاكهة.[6][7] ربما تعتبر تلك التقارير العمل الريادي في مجال ترسيب الكروماتين مناعيا. بعدها قام إلكسندر فارشافسكي ومساعديه بتطوير وتعديل XChIP اللذين درسوا توزع الهيستون H4 على جين الصدمة الحرارية الارتباط المتصالب بالفورم ألدهيد بعدها تم تطوير وتعديل هذه الطريقة.[8][9][10] وصفت طريقة الـNChIP أول مرة من قبل Hebbes وزملائه عام 1988 وتم تطويرها وتعديلها بسرعة.[11][12] تحتاج ChIP النموذجية أربعة إلى خمسة أيام و 106~ 107 خلايا على الاقل. الان هناك طرق جديدة للـChIPيمكن انجازها بعدد أقل من الخلايا حوالي 100~1000 وبيوم واحد.
- (Carrier ChIP (CChIP
يمكن استخدام عدد فليل من الخلايا حوال 100 خلية من خلال إضافة خلايا Drosophila كحامل للكروماتين وذلك لتقليل خسارة الكروماتين الهدف وتسهيل عملية ترسيبه ولكنها تحتاج مشارع primers ذات نوعية عالية لتحديد كروماتين الخلية الهدف وتمييزه عن الكروماتين الحامل الغريب وتحتاج يومين إلى ثلاثة ايام.[13]
- (Fast ChIP (qChIP
تقلل هذه الطريقة من الوقت اللازم لاجراء المقايسة مقارنة بالـChIP التقليدية من خلال اختصار خطوتان:
- حمام صوتي يسرع ارتباط الضد مع البروتين الهدف مما ينقص زمن الترسيب المناعي.
- استخدام اجراء عزل الـDNA بالاعتماد على الراتنج (Chelex-100) يقلل زمن الارتباط التصالب العكسي وعزل الـDNA.
لكن هذا البروتوكول السريع مناسب فقط عندما يكون عدد خلايا العينة كبير (حوالي106~ 107)[14][15] تخضع عينات الكروماتين المجزأ إلى أكثر من 24 جزء الىPCR-ready DNA خلال 5 ساعات مما يسمح بأن تكون عوامل الكروماتين المتضاعفة مسابير خلال نقاط زمنية متعددة.[16]
- (Quick and quantitative ChIP (Q2ChIP
تستخدم هذه المقايسة 100,000 خلية كمواد بدء وهي مناسبة لاجراء ترسيب المناعي للكروماتين لأكثر من 100 هيستونأو 100 عامل انتساخ لذا يمكن تحضي العديد من عينات الكروماتين بشكل متوازي وحفظها وتجرى خلال يوم.[17]
- (MicroChIP (µChIP
يحضر الكروماتين من 1,000 خلية ويمكن اجراء أكثر من 8 عمليات ترسيب منعي للكروماتين بشكل متوازي بدون حامل يمكن البدء بهذه المقايسة بـ 100 خلية لكنها مناسبة لاجراء ترسيب مناعي واحد ويمكن استخدام خزعة نسيجية صغيرة (1 mm3) . يتم اجراء هذا النوع خلال يوم.[18][19]
- Matrix ChIP
تعتبر مقايسة ترسيب مناعي للكروماتين معتمدة على الصفيحة المكروية -based ChIP assaymicroplate حيث تتم جميع الخطوات ضمن ابار صفيحة مكروية بدون نقل العينة تسمح هذه الطريقة باجراء 96 ترسيب مناعي للكروماتين للهيستون وبروتينات متعددة مرتبطة بالـDNA خلال يوم.[20] كما تم تطبيق الترسيب المنعي للكروماتين لتحليل واسع للجينوم من خلال ربطه بتفنية الـmicroarray(ChIP-on-chip) أو تقنية الجيل الثاني من تسلسل الـDNA(Chip-Sequencing) كما يمكن ربطها بتسلسل النهاية الموسومة المتزاوجة paired-end tags sequencingفي تحليل تداخل الكروماتين باستخدام تقنية بتسلسل النهاية الموسومة المتزاوجة (ChIA-PET).[21][22][23]
محدودية الترسيب المنعي للكروماتين
- مقايسة الكميات الكبيرة باستخدام كائنات حية سليمة لانه يجب تشكيل أضداد لكل عامل انتساخ أو يجب ان يعبر الكائن الحي عن محدد مستضدي موسوم بعامل الانتساخ ضروري للهذا الاجراء.
- تواجه الدراسات البحثية لانماط التعبير الجيني المتباين في الكائنات الصغيرة مشاكل وذلك لان التعبير الجيني يكون بمستويات قليلة وفي عدد خلايا قليل وخلال وقت ضيق.
- لاتستطيع تجارب الترسيب المنعي للكروماتين تمييز الاشكال الفراغية isoform لعوامل النتساخ المختلفة (الشكل الفراغي للبروتين بروتين نظير).
انظر أيضاً
- W:RIP-Chip ،وهي تقنية مشابهة لتحليل تفاعلات الحمض النووي الريبي RNA مع البروتين.
- W:DamID، تقنية بديلة لرسم خرائط المواقع لا تتطلب أجسام مضادة محددة.
- W:ChIP-exo،وهي تقنية تضيف معالجة بأنزيم إكسو نوكلياز لعملية الترسيب المناعي للكروماتين لتحصل على ما يصل إلى زوج أساس مفرد في مواقع الربط
- W:ChIP-on-chip، يجمع بين الترسيب المناعي للكروماتين مع تقنية المصفوفة الرقيقة microarray .
المراجع
- Collas, Philippe. (January 2010). "The Current State of Chromatin Immunoprecipitation". Molecular Biotechnology 45 (1): 87–100. doi:10.1007/s12033-009-9239-8. ببمد 20077036.
- Gilmour DS, Lis JT (August 1985). "In vivo interactions of RNA polymerase II with genes of Drosophila melanogaster". Molecular and Cellular Biology 5 (8): 2009–18. PMC 366919. ببمد 3018544. Retrieved 2010-03-13.
- Jackson, Vaughn (November 1978). "Studies on histone organization in the nucleosome using formaldehyde as a reversible cross-linking agent". Cell 15 (3): 945–54. doi:10.1016/0092-8674(78)90278-7. ببمد 569554. Retrieved 2010-03-13.
- Bauer UM, Daujat S, Nielsen SJ, Nightingale K, Kouzarides T (January 2002). "Methylation at arginine 17 of histone H3 is linked to gene activation". EMBO Reports 3 (1): 39–44. doi:10.1093/embo-reports/kvf013. PMC 1083932. ببمد 11751582. |accessdate= requires |url= (help)
- Viens A et al. "Use of protein biotinylation in vivo for chromatin immunoprecipitation" (2004) Analytical Biochemistry 325(1):68-76 [1]
- [2]
- [3]
- Varshavsky A (December 2008). "Discovery of cellular regulation by protein degradation". Journal of Biological Chemistry 283 (50): 34469–89. doi:10.1074/jbc.X800009200. ببمد 18708349. Retrieved 2010-03-06.
- Solomon, Mark J; Larsen Pamela L; Varshavsky, Alexander. (June 1988). "Mapping protein-DNA interactions in vivo with formaldehyde: evidence that histone H4 is retained on a highly transcribed gene". Cell 53 (6): 937–47. doi:10.1016/S0092-8674(88)90469-2. ببمد 2454748. Retrieved 2010-03-06.
- Orlando V (March 2000). "Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation". Trends in Biochemical Sciences 25 (3): 99–104. doi:10.1016/S0968-0004(99)01535-2. ببمد 10694875. Retrieved 2010-03-14.
- Hebbes, Tim R; Thorne, Alan W; Crane-Robinson C. (May 1988). "A direct link between core histone acetylation and transcriptionally active chromatin". The EMBO Journal 7 (5): 1395–402. PMC 458389. ببمد 3409869.
- O'Neill, Laura P; Turner, Bryan M (September 2003). "Immunoprecipitation of native chromatin: NChIP". Methods (San Diego, Calif.) 31 (1): 76–82. doi:10.1016/S1046-2023(03)00090-2. ببمد 12893176. Retrieved 2010-03-14.
- O'Neill, Laura P; VerMilyea, Matthew D; Turner, Bryan M (July 2006). "Epigenetic characterization of the early embryo with a chromatin immunoprecipitation protocol applicable to small cell populations". Nature Genetics 38 (7): 835–41. doi:10.1038/ng1820. ببمد 16767102.
- Nelson, Joel D; Denisenko, Oleg; Sova, Pavel; Bomsztyk, Karol (2006). "Fast chromatin immunoprecipitation assay". Nucleic Acids Research 34 (1): e2. doi:10.1093/nar/gnj004. PMC 1325209. ببمد 16397291.
- ^ Nelson, Joel D; Denisenko, Oleg; Bomsztyk, Karol (2006). "Protocol for the fast chromatin immunoprecipitation (ChIP) method". Nature Protocols 1 (1): 179–85. doi:10.1038/nprot.2006.27. ببمد 17406230.
- Nelson J, Denisenko O, Bomsztyk K (2009). "The fast chromatin immunoprecipitation method". Methods in Molecular Biology 567: 45–57. doi:10.1007/978-1-60327-414-2_3. ببمد 19588084.
- Dahl, John Arne; Collas, Philippe (April 2007). "Q2ChIP, a quick and quantitative chromatin immunoprecipitation assay, unravels epigenetic dynamics of developmentally regulated genes in human carcinoma cells". Stem Cells 25 (4): 1037–46. doi:10.1634/stemcells.2006-0430. ببمد 17272500.
- Dahl, John Arne; Collas, Philippe (2008). "A rapid micro chromatin immunoprecipitation assay (microChIP)". Nature Protocols 3 (6): 1032–45. doi:10.1038/nprot.2008.68. ببمد 18536650.
- Dahl, John Arne; Collas, Philippe (2009). "MicroChIP: chromatin immunoprecipitation for small cell numbers". Methods in Molecular Biology 567: 59–74. doi:10.1007/978-1-60327-414-2_4. ببمد 19588085.
- Flanagin, Steve ; Nelson, Joel D; Castner, David G; Denisenko, Oleg; Bomsztyk, Karol (February 2008). "Microplate-based chromatin immunoprecipitation method, Matrix ChIP: a platform to study signaling of complex genomic events". Nucleic Acids Research 36 (3): e17. doi:10.1093/nar/gkn001. PMC 2241906. ببمد 18203739.
- Fullwood, Melissa J; Han, Yuyuan; Wei, Chia-Lin; Ruan, Xiaoan; Ruan, Yijun (January 2010). "Chromatin interaction analysis using paired-end tag sequencing". Current Protocols in Molecular Biology. Chapter 21: Unit 21.15.1–25. doi:10.1002/0471142727.mb2115s89. ببمد 20069536.
- Li, Guoliang; Fullwood, Melissa J; Xu, Han; Mulawadi, Fabianus Hendriyan; Velkov, Stoyan; Vega, Vinsensius; Ariyaratne, Pramila Nuwantha; Mohamed, Yusoff Bin; Ooi, Hong-Sain; Tennakoon, Chandana; Wei, Chia-Lin; Ruan, Yijun; Sung, Wing-Kin (February 2010). "ChIA-PET tool for comprehensive chromatin interaction analysis with paired-end tag sequencing". Genome Biology 11 (2): R22. doi:10.1186/gb-2010-11-2-r22. PMC 2872882. ببمد 20181287.
- "ChIA-PET: Novel Method For 3-D Whole Genome Mapping Research". ScienceDaily. Agency for Science, Technology and Research (A*STAR), Singapore. 2009-11-08. Retrieved 14 March 2010.
روابط أخرى
- Chromatin immunoprecipitation في المَكتبة الوَطنية الأمريكية للطب نظام فهرسة المواضيع الطبية (MeSH).
- EpigenomeNOE.com
- Chromatin Immunopreciptation (ChIP) on Unfixed Chromatin from Cells and Tissues to Analyze Histone Modifications
- Chromatin Immunoprecipitation (ChIP) of Protein Complexes: Mapping of Genomic Targets of Nuclear Proteins in Cultured Cells
- بوابة علم الأحياء الخلوي والجزيئي
