تصاحب جيني
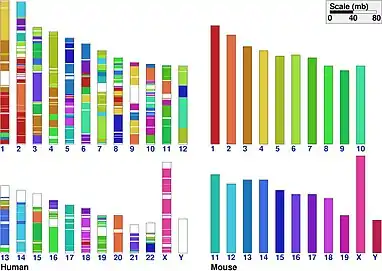
في علم الوراثة الكلاسيكي , يعرف التصاحب الجيني أو التزامن، على انه التوطين المادي المشترك للمكان الجيني على نفس الكروموسوم داخل الفرد أو الفصيلة. اليوم، علماء الوراثة يشيرون إلى التصاحب الجيني عادة باعتباره التحفظ على كتل النظام داخل مجموعتين من الكروموسومات التي يتم مقارنتها مع بعضها البعض.
يرتبط المفهوم الكلاسيكي بالارتباط الجيني من حين فكرة ان انشاء الروابط بين موقعي كروموسومات يتم من خلال مراقبة ترددات إعادة التركيب أقل من المتوقع بينهما. على النقيض من ذلك، فإن أي مكان على نفس الكروموسوم يعرف بالتصاحب الجيني (التزامن) ، حتى إذا كان لا يمكن تمييز تردد إعادة تركيبه عن الموقع غير المرتبط من خلال التجارب العملية. و بالتالي، من الناحية النظرية، تكون جميع المواقع المرتبطة متزامنة، ولكن ليس كل المواقع المتزامنة مرتبطة بالضرورة. وبالمثل، في علم الجينوم ، فإن الموقع الجيني على الكروموسوم متزامن بغض النظر عما إذا كان يمكن إنشاء هذه العلاقة من خلال طرق تجريبية مثل تسلسل / تجميع الحمض النووي ، أو المشي الجينومي، أو التوطين الجسدي أو رسم "خرائط الهاب".
يستخدم طلاب علم الوراثة مصطلح متزامنة لوصف الحالة التي تم فيها تعيين موقعين جينيين للكروموسوم نفسه ولكن لا يزال من الممكن فصلهما بمسافة كبيرة بما فيه الكفاية في وحدات الخريطة التي لم يتم إثبات الارتباط الوراثي فيها.
تقدم موسوعة بريتانكا (Britanca) الوصف الأتي للتزامن:
لقد أتاح التسلسل الجيني ورسم الخرائط مقارنة بين الهياكل العامة للجينوم للعديد من الأنواع المختلفة. الاستنتاج العام هو أن الكائنات الحية ذات الاختلاف الحديث نسبيًا تُظهر كتلًا متشابهة من الجينات في المواضع النسبية نفسها في الجينوم. تسمى هذه الحالة التزامن، وتُترجم تقريبًا على أنها تمتلك تسلسلات كروموسوم مشتركة. على سبيل المثال، العديد من جينات البشر متزامنة مع جينات الثدييات الأخرى. ليس فقط القرود ولكن أيضًا الأبقار والفئران وما إلى ذلك. يمكن أن توضح دراسة التوافق النشط كيفية قطع الجينوم ولصقه في سياق التطور.
في علم أصول الكلمات
التصاحب الجيني أو التزامن هو مصطلح حديث "يعني على نفس الشريط" ؛
و في اليونانية :
σύν ، syn = مع + ταινία ، tainiā = النطاق
في إشارة إلى نفس ترتيب الجينات في سلسلتين (متماثلتين) من الحمض النووي (أو الكروموسومات).
التصاحب الجيني المشترك
يصف التصاحب الجيني المشترك (المعروف أيضًا باسم التزامن المحفوظ) التوطين المشترك المحفوظ للجينات على الكروموسومات من الأنواع المختلفة. أثناء التطور، قد تؤدي إعادة الترتيب إلى الجينوم مثل انتقال الكروموسومات إلى فصل موقعين، مما يؤدي إلى فقدان التزامن بينهما. على العكس من ذلك، يمكن أن تنقل الانتقالات قطعتين منفصلتين سابقًا من الكروموسومات معًا، مما يؤدي إلى اكتساب التزامن بين المواقع. يمكن للتزامن المشترك الأقوى من المتوقع أن يعكس الاختيار للعلاقات الوظيفية بين الجينات المتزامنة، مثل مجموعات الأليات التي تكون مفيدة عندما تُورث معًا، أو الآليات التنظيمية المشتركة.[1]
يستخدم المصطلح أحيانًا أيضًا لوصف عملية الحفاظ على الترتيب الدقيق للجينات على كروموسوم ينتقل من سلف مشترك.[2][3][4][5] على الرغم من رفض العديد من علماء الوراثة استخدام هذا المصطلح.[6]
إن تحليل التزامن من منطلق ترتيب الجينات له عدة تطبيقات في علم الجينوم. التصاحب الجيني المشترك هو أحد المعايير الأكثر موثوقية لتأسيس علم تركيب المناطق الجينومية في الأنواع المختلفة. بالإضافة إلى ذلك، يمكن للحفظ الاستثنائي للتزامن أن يعكس العلاقات الوظيفية الهامة بين الجينات.
على سبيل المثال، يتم الحفاظ على ترتيب الجينات في عنقودية النحت (Hox cluster)
وهي المحددات الرئيسية لخطة جسم الحيوان والتي تتفاعل مع بعضها البعض بطرق حرجة، وبشكل أساسي في جميع أنحاء مملكة الحيوان.[7]
يستخدم التصاحب الجيني على نطاق واسع في دراسة الجينوم المعقد، حيث يسمح الجينوم المقارن بوجود الجينات وربما وظيفتها في كائن نموذجي أبسط لاستنتاج تلك الموجودة في كائن أكثر تعقيدًا. على سبيل المثال، يحتوي القمح على جينوم كبير ومعقد للغاية يصعب دراسته. في عام 1994 أظهر البحث الذي أجراه مركز جون إينيس في إنجلترا والمعهد الوطني للأبحاث الزراعية في اليابان أن جينوم الأرز الأصغر كان له بنية وراثية مشابهة لتلك الموجودة في القمح. [8] وجدت دراسة أخرى أن العديد من الحبوب متزامنة [9] وبالتالي يمكن استخدام نباتات مثل الأرز أو العشب كنموذج للعثور على الجينات أو العلامات الجينية ذات الأهمية التي يمكن استخدامها في تربية القمح والبحث. في هذا السياق، كان الترادف ضروريًا أيضًا في تحديد منطقة مهمة للغاية في القمح، وهو موقع PH1 الذي يشارك في استقرار الجينوم والخصوبة، والذي تم تحديده باستخدام معلومات من المناطق المتزامنة في الأرز والعشب.[10]
يستخدم التصاحب الجيني أيضًا على نطاق واسع في علم الجينوم الميكروبي. في المستجذريات والأمعائيات، تشفر الجينات المتزامنة عددًا كبيرًا من وظائف الخلية الأساسية وتمثل مستوى عالٍ من العلاقات الوظيفية.[11]
يمكن أيضًا استخدام أنماط الفواصل المتزامنة أو الفواصل المتداخلة كأحرف لاستدلال العلاقات الجينية بين عدة أنواع، وحتى لاستنتاج تنظيم الجينوم لأنواع الأسلاف المنقرضة. يتم أحيانًا التمييز بين النوعي بين الحَكَم العظمي، والحفاظ على التشابه في أجزاء كبيرة من الكروموسوم، والحَقْنُ العَضَلِيّ، والحفاظ على التشابه لبضعة جينات فقط في كل مرة.
الكشف عن طريق الحساب
يمكن استنتاج التوافق المشترك بين الأنواع المختلفة من تسلسلها الجينومي. يتم ذلك عادة باستخدام نسخة من خوارزمية MCScan ، التي تجد كتل متزامنة بين الأنواع من خلال مقارنة جيناتها المتماثلة والبحث عن أنماط مشتركة متداخلة مشتركة على مقياس كروموسومي أو مترابط. عادة ما يتم تحديد التماثلات على أساس درجات بت عالية التي تحدث بين الجينومات المتعددة. من هنا، يتم استخدام البرمجة الديناميكية لتحديد أفضل مسار لتسجيل الجينات المتجانسة المشتركة بين الأنواع، مع الأخذ بعين الاعتبار فقدان الجينات المحتمل والكسب الذي ربما حدث في التاريخ التطوري للنوع.[12]
مراجع
- "Transcription unit conservation in the three domains of life: a perspective from Escherichia coli". Trends in Genetics. 17 (4): 175–177. 2001. doi:10.1016/S0168-9525(01)02241-7. PMID 11275307. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Genomic regulatory blocks underlie extensive microsynteny conservation in insects". Genome Res. 17 (12): 1898–908. 2007. doi:10.1101/gr.6669607. PMC 2099597. PMID 17989259. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Evolutionary rate analyses of orthologs and paralogs from 12 Drosophila genomes". Genome Res. 17 (12): 1837–49. 2007. doi:10.1101/gr.6249707. PMC 2099592. PMID 17989258. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "The determinants of gene order conservation in yeasts". Genome Biol. 8 (11): R233. 2007. doi:10.1186/gb-2007-8-11-r233. PMC 2258174. PMID 17983469. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Gene order and recombination rate in homologous chromosome regions of the chicken and a passerine bird". Mol. Biol. Evol. 24 (7): 1537–52. 2007. doi:10.1093/molbev/msm071. PMID 17434902. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Incorrect use of the term synteny". Nature Genetics. 23 (4): 387. December 1999. doi:10.1038/70486. PMID 10581019. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Zebrafish hox clusters and vertebrate genome evolution". Science. 282 (5394): 1711–4. November 1998. doi:10.1126/science.282.5394.1711. PMID 9831563. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Conservation of genome structure between rice and wheat". Nature Biotechnology. 12 (3): 276–278. 1994. doi:10.1038/nbt0394-276. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Cereal genome evolution. Grasses, line up and form a circle". Current Biology. 5 (7): 737–9. July 1995. doi:10.1016/S0960-9822(95)00148-5. PMID 7583118. الوسيط
|CitationClass=تم تجاهله (مساعدة) - "Molecular characterization of Ph1 as a major chromosome pairing locus in polyploid wheat". Nature. 439 (7077): 749–52. February 2006. doi:10.1038/nature04434. PMID 16467840. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Guerrero, G; Peralta, H; Aguilar, A; Díaz, R; Villalobos, MA; Medrano-Soto, A; Mora, J (17 October 2005). "Evolutionary, structural and functional relationships revealed by comparative analysis of syntenic genes in Rhizobiales". BMC Evolutionary Biology. 5: 55. doi:10.1186/1471-2148-5-55. PMC 1276791. PMID 16229745. الوسيط
|CitationClass=تم تجاهله (مساعدة) - Wang, Y; Tang, H; Debarry, JD; Tan, X; Li, J; Wang, X; Lee, TH; Jin, H; Marler, B; Guo, H; Kissinger, JC; Paterson, AH (April 2012). "MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity". Nucleic Acids Research. 40 (7): e49. doi:10.1093/nar/gkr1293. PMC 3326336. PMID 22217600. الوسيط
|CitationClass=تم تجاهله (مساعدة)
- بوابة علم الأحياء الخلوي والجزيئي